2.Blast
1) Pipeline
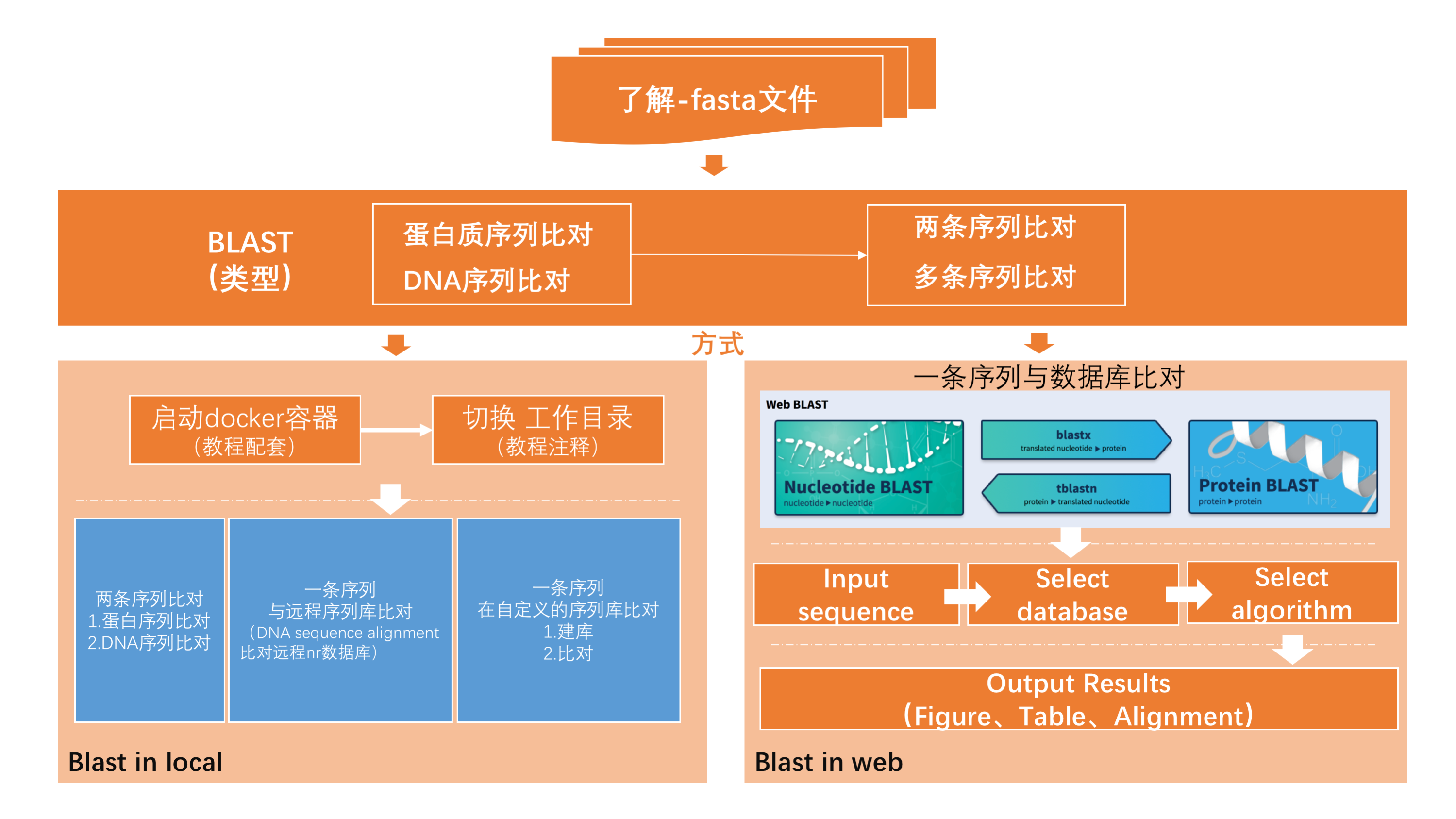
2) Data Structure
了解存储sequence的常用文件格式
FASTA格式 (.fasta or .fa )
>gi|47115317|emb|CAG28618.1| VIM [Homo sapiens] MSTRSVSSSSYRRMFGGPGTASRPSSSRSYVTTSTRTYSLGSALRPSTSRSLYASSPGGVYATRSSAVRL
The word following the ">" symbol is the identifier of the sequence, and the rest of the line is the description (optional). Normally, identifiers are simply protein accession, name or Entrez gi's (e.g., Q5I7T1, AG10B_HUMAN, 129295), but a bar-separated NCBI sequence identifier (e.g., gi|129295) will also be accepted. Any arbitrary user-specified sequence identifier can also be used (e.g., CLONE00073452).
2a) Inputs
| Format | Description | Notes |
|---|---|---|
| fasta | contains gene name and sequence | gene name start with > |
2b) Outputs
| Format | Description | Notes |
|---|---|---|
| blastp | query aligned to database | - |
3) Blast in local
(1) 准备
进入到容器(在自己电脑的 Terminal 中运行,详情请参见 这里):
docker ps # List containers
| CONTAINER ID | IMAGE | COMMAND | CREATED | STATUS | PORTS | NAMES |
|---|---|---|---|---|---|---|
| 4b18158cd00b | bioinfo_tsinghua | "/bin/bash" | 6 days ago | Up 7 hours | -- | bioinfo_tsinghua |
docker exec -it bioinfo_tsinghua bash
# docker 命令中 bioinfo_tsinghua 是 container 的名字。
如果 docker ps 没有显示 bioinfo_tsinghua 正在运行,启动 bioinfo_tsinghua container.
docker run --name=bioinfo_tsinghua -dt --restart unless-stopped -v ~/Desktop/bioinfo_tsinghua_share:/home/test/share bioinfo_tsinghua
docker ps # List containers
docker exec -it bioinfo_tsinghua bash
以下步骤均在 /home/test/blast/ 下进行:
cd /home/test/blast/
准备输出目录
mkdir output
(2) 两条序列比对
Protein sequence alignment
利用blastp进行蛋白质比对
blastp -query protein/VIM.fasta -subject protein/NMD.fasta -out output/blastp
VIM.fasta 与 NMD.fasta 分别是金属beta酶家族的两个亚种酶的序列
DNA sequence alignment
利用blastn进行DNA序列比对
blastn -query dna/H1N1-HA.fasta -subject dna/H7N9-HA.fasta -out output/blastn
H1N1-HA.fasta 与H7N9-HA.fasta 是流感病毒序列文件
(3) 一条序列与远程序列库比对
**_Protein sequence alignment 比对远程pdb数据库**_
运行时需要联网,无法上网请跳过。
blastp -query protein/VIM.fasta -db pdb -remote -out output/blastp_remote
DNA sequence alignment 比对远程nr数据库
运行时需要联网,无法上网请跳过。
blastn -query dna/H1N1-HA.fasta -db nr -remote -out output/blastn_remote
(4) 一条序列在自定义的序列库里比对
例如:在yeast基因组序列中搜索Yeast.fasta序列
Step 1: 建库
makeblastdb -dbtype nucl -in dna/YeastGenome.fa -out database/YeastGenome
-dbtype: 待建库的类型(nucl,prot)-in: 待建库的序列文件-out: 序列库名前缀
Step 2: 比对
blastn -query dna/Yeast.fasta -db database/YeastGenome -out output/Yeast.blastn
4) Blast online
首先,进入NCBI官网在线BLAST,显示如下图所示界面,选择一种序列比对类型,然后根据提示进行序列比对。
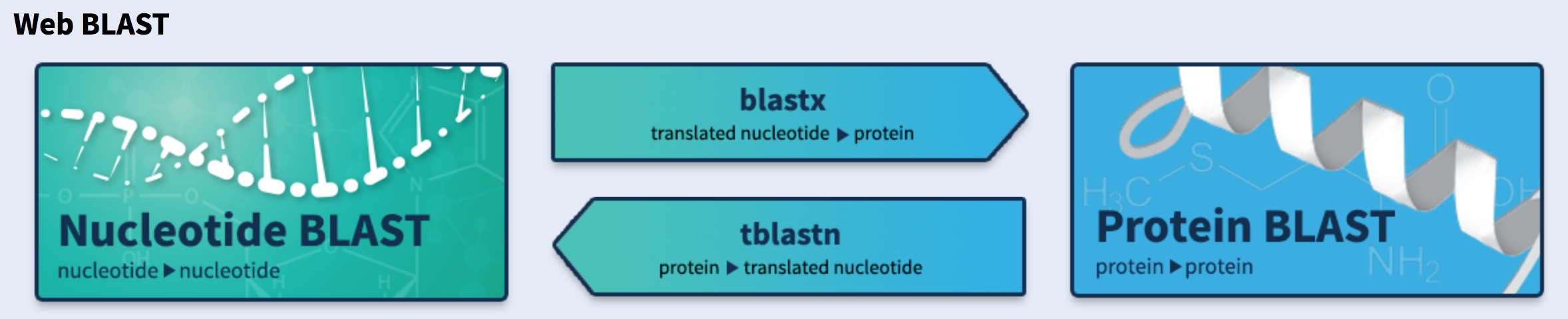
Running Steps
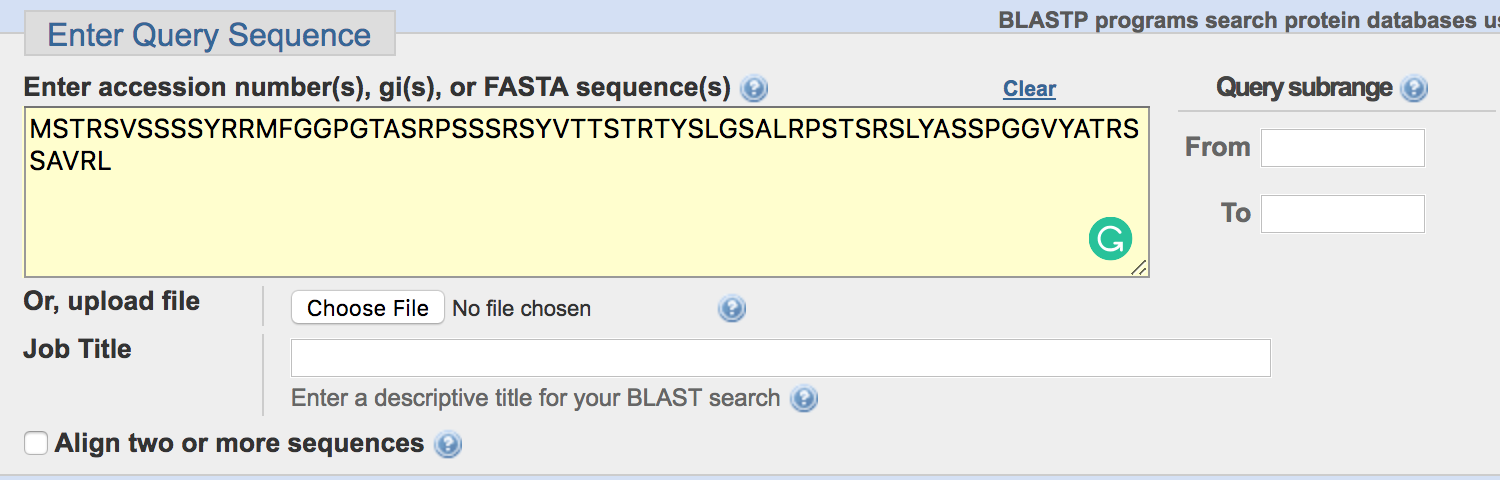
(1) Input sequence
MSTRSVSSSSYRRMFGGPGTASRPSSSRSYVTTSTRTYSLGSALRPSTSRSLYASSPGGVYATRSSAVRL
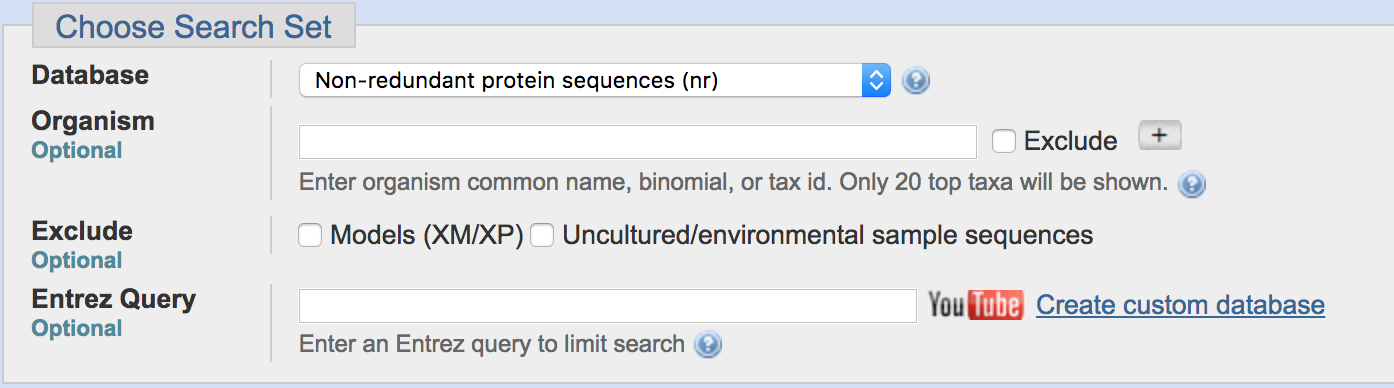
(2) Select database
(3) Select algorithm

(4) Output results
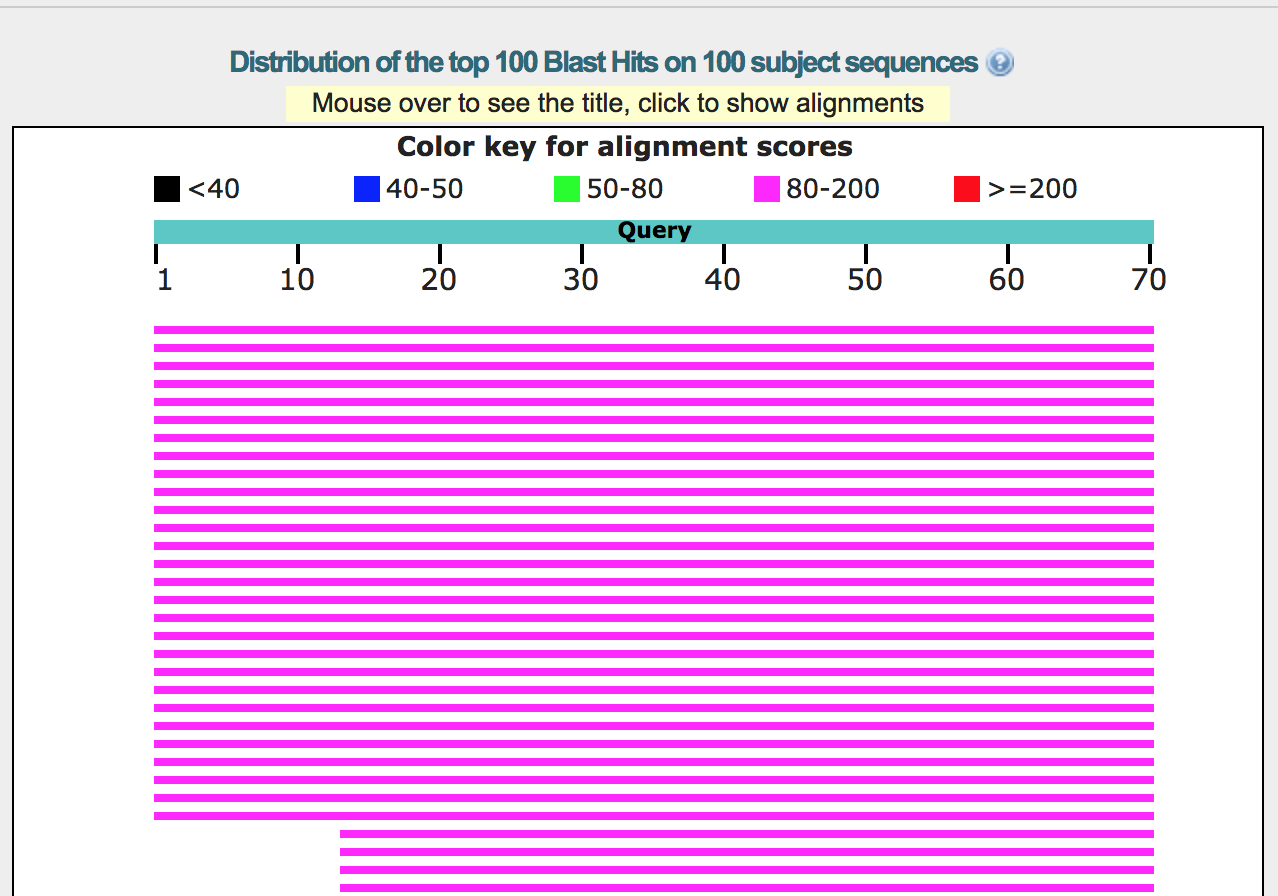
(5) Output table
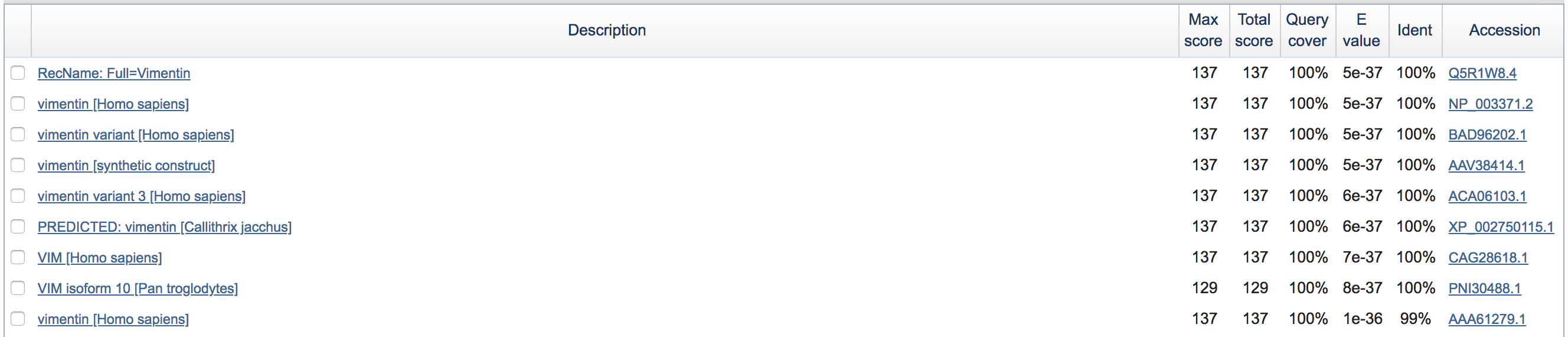
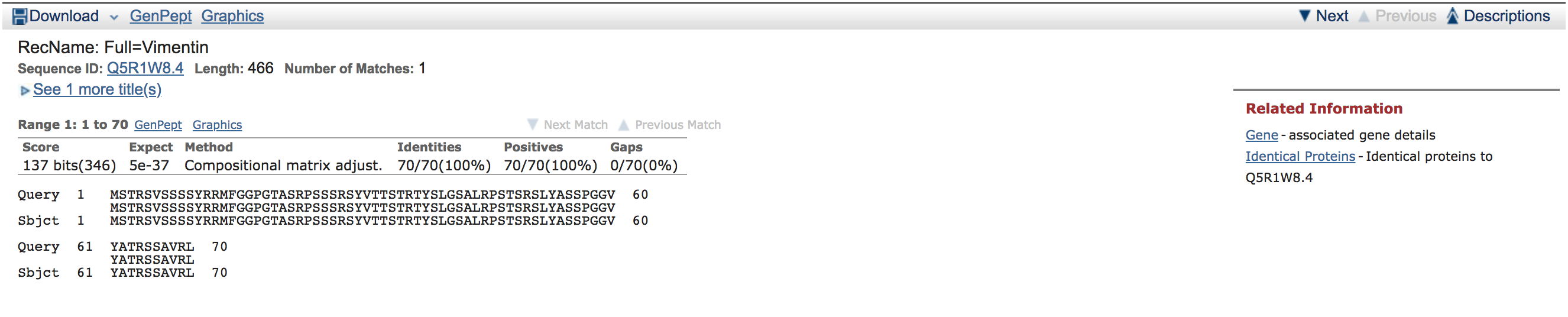
(6) Alignment details
5) Tips/Utilities
(1) 查看序列比对结果文件的方式(blast in local)
方式1-利用docker挂载电脑主机目录,同步文件(该方法详见Appendix iii. docker章节tips/utilities部分)。
方式2-利用vi文本编辑工具查看(该方法详见HUXI Notes_Linux 章节tips/utilities-7.vi文件编辑器使用部分 )。
(2) A better view of fasta file
less -S dna/H1N1-HA.fasta # chop long lines rather than wrap them (记得按 q 退出)
(3) 安装blast及其子程序blastn, blastp,blastx, ...
Note: Docker 中已经装好
安装方式1-自动安装(推荐方式):
Ubuntu 自动安装软件方法: sudo apt-get install 软件名称
这里 blast 由 ncbi-blast+ 提供
安装方式2-手动安装:
寻找类似如下文件:
a. 32位计算机(老机器)安装文件:如 ncbi-blast-2.2.28+-ia32-linux.tar.gz
b. 64位机器安装文件: 如 ncbi-blast-2.2.28+-x64-linux.tar.gz
(可以通过 uname -a 查看机器类型是64还是32位)
6) Homework and more
(1) Blast sequence with mouse genome.
请使用网页版的 blastp, 将上面的蛋白序列只与 mouse protein database 进行比对, 设置输出结果最多保留10个, E 值最大为 0.5。将操作过程和结果截图。
休息一会
Jim Kent

Jim Kent 1960年生于夏威夷,在旧金山长大。
Kent的编程生涯始于23岁,1983年Kent开始在Island Graphic Inc工作。当时Kent为Amiga家用电脑编写了一个能够结合3D渐变效果与2D简单动画的软件。
1985年他干脆自己开了一家软件公司 Dancing Flame, 将之前自己写的动画程序创建成为一个汇集各种动画和绘画功能的程序Cyber Paint,为CAD-3D开发提供便利。这是是第一个允许用户跨时间制作压缩视频的软件。之后他还开发了软件Autodesk Animator用于个人电脑中,可以为各种视频游戏创作艺术作品。
2000年Kent在攻读加州大学圣克鲁兹生物学博士学位,此时人类基因组计划进行到后期,他写出了GigAssembler, 帮助人类基因组项目能够拼装和发布人类基因组序列。
在GigAssembler之后,Kent继续编写BLAT(BLAST-like alignment tool)和参与维护UCSC Genome Browser 来帮助分析重要的基因组数据。目前,Kent仍然在UCSC工作,主要是在网络工具上帮助理解人类基因组。他帮助维护和升级浏览器,并致力于比较基因组学, Parasol,UCSC kilocluster的运营和ENCODE项目。